分子植物育种
(
网络版
), 2016
年
,
第
14
卷
,
第
1008
-
1015
页
Fenzi Zhiwu Yuzhong (Online), 2016, Vol.14, 1008
-
1015
1010
表
2
测序数据的组分构成
Table 2 The composition and ratios of clean data
样品
Sample
总碱基数
Total base
叶绿体
Cp
线粒体
Mt
核
Nuclear
碱基数
Base
比率
(%)
Ratio (%)
测序深度
Depth
碱基数
Base
比率
(%)
Ratio (%)
测序深度
Depth
碱基数
Base
比率
(%)
Ratio (%)
测序深度
Depth
RS41
244 345 283
123 516 541
50.55
823.44
28 539 529
11.68
119.41
92 289 213
37.77
0.18
RS42
243 804 470
131 288 707
53.85
875.26
26 964 774
11.06
112.82
85 550 989
35.09
0.17
RS43
258 782 888
129 236 174
49.94
861.57
27 301 595
10.55
114.23
102 245 119
39.51
0.20
RS44
256 459 385
126 024 142
49.14
840.16
31 416 275
12.25
131.45
99 018 969
38.61
0.20
RS45
254 698 444
134 098 731
52.65
893.99
25 775 483
10.12
107.85
94 824 231
37.23
0.19
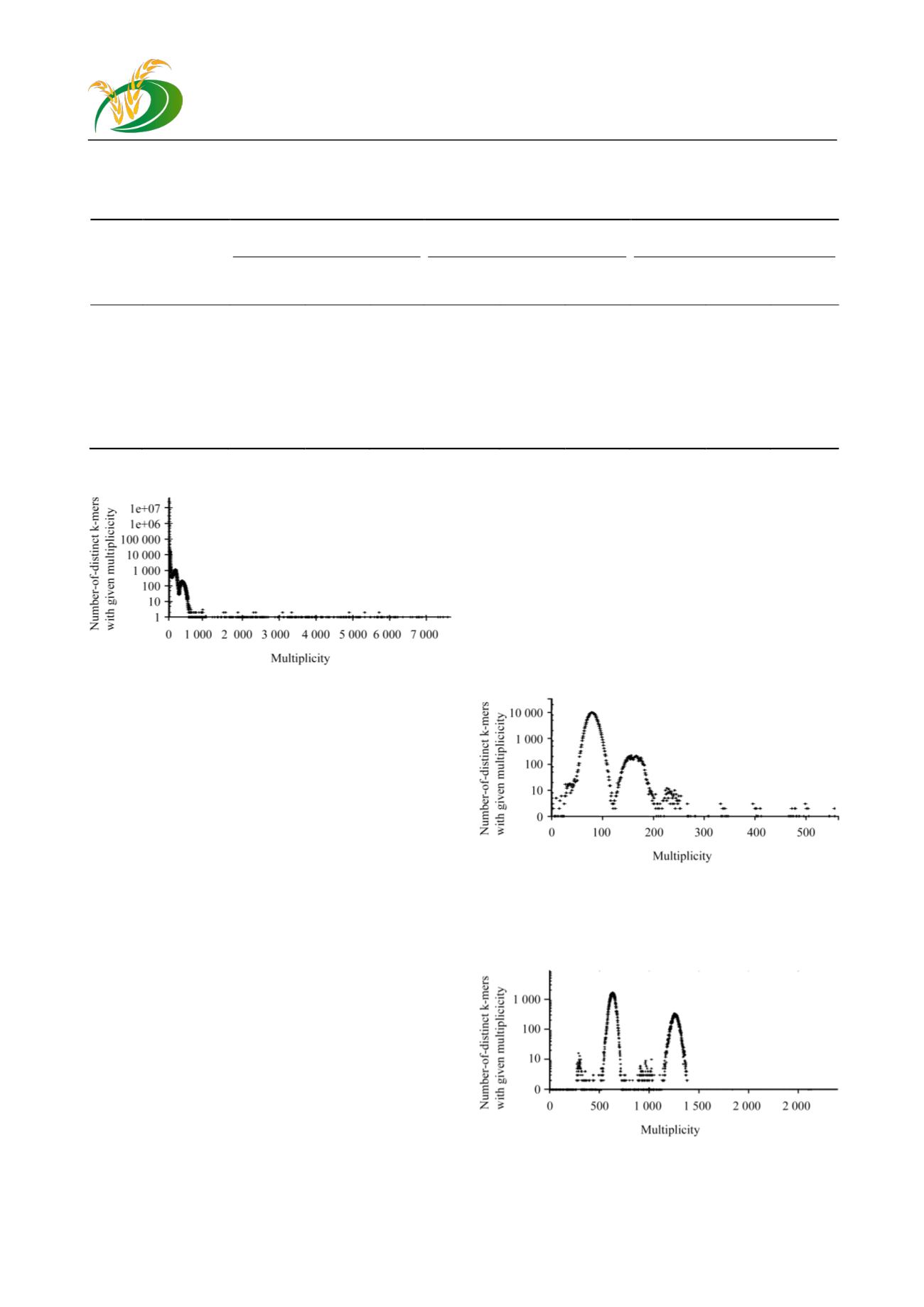
图
1 RS41
样品原始数据
Kmer
频度分布
Figure 1 Kmer frequency distribution of RS41’s raw data
经软件过滤后,对
Reads
进行
21 mer
的
Kmer
分析可见明显的高频度组分,以
41
样品为例:叶
绿体
Reads
,
Kmer
分析表明呈现两个明显的尖锐的
峰
(
图
2)
,即该数据存在两个组分,针对第
1
个峰计
算的测序深度为,第二个峰计算的测序深度为,接
近第
1
个组分的两倍,推测是
1
个重复序列。从数
据量上看,第一组分中占有的比例是
6/8,
大约占有
碱基量
92 637 405
,该数值除测序深度
820
可得该
组分分子量为
110 k
。第二组份占有的比例是
2/8
,
大约占有碱基数是
30 879 135
,该数值除测序深
度
1 600
,得该组分子量约为
20 k
,合计分子量为
该数值符合预期的萝卜叶绿体基因组碱基数。针对
线粒体组装的
Reads
,其
Kmer
分布同样呈现明显
的双峰
(
图
3)
,比例关系为
(26:1)
,利用同样的算
法,估测的对应分子量分别为
221 k
及
9 k
,合计
估测线粒体基因组为
239 k
。
其余
42
、
43
、
44
、
45
四个样品进行的
21mer
,
Kmer
频度分析结果均与
41
号样品相似。预测的线
粒体基因组
240~250 k
,预测的叶绿体基因组
140~150 k
,均符合预期。
Kmer
分析表明,通过
BWA
比对及软件筛选搜
集了来自不同细胞器的
Reads
。原始数据的分类过
滤,有效提高了
Reads
的专一性。这为后续的组装
打下了基础。
图
2 RS41
样品数据过滤后叶绿体数据
Kmer
频度分布
Figure 2 Kmer frequency distribution of filtered RS41’s
chloroplast data
图
3 RS41
样品数据过滤后叶绿体数据
Kmer
频度分布
Figure 3 Kmer frequency distribution of filtered RS41’s
mitochondrial data


