计算分子生物学
(
网络版
)
Jisuan Fenzi Shengwuxue (Online)
Copyright © 2015 BioPublisher Jisuan Fenzi Shengwuxue | Vol.4 | No.1 | 1–12
如果
GFF
文档已经包含
FASTA
格式数据的情
况下,
FASTA
格式的蛋白质序列文档就是可选择
项。
ProteoAnnotator
需要用户上传一个套基因组坐
标
(genomic coordinates)
和蛋白质序列作为物种基
因组的正式注释模式,并被标记为
“A”
套基因
/
蛋白
质以用作进一步的分析。在某些没有正式基因组模
式的测序基因组,用户应当上传一套最好质量的比
如由基因预测软件预测过的基因模式,接着用户可
自行决定是否按照预测基因模式质量顺序为参考
上传其他的并依次标注为“
B
”,“
C
”,“
D
”等等。
ProteoAnnotator
在创建诱饵数据用于
SearchGUI
和
预处理与后处理过程中,及在使用
Omssa
和
X!Tandem
进行
MS/MS
搜索中都使用了
MzidLib
(Ghali et al., 2013)
。
SearchGUI
作为一个开放源工具
可以在一个质谱搜索中使用不同的开放源代码的
搜索引擎
(Omssa, X!Tandem, MSGF+
和
MS-Amanda)
(Vaudel et al., 2011)
。
ProteoAnnotaotr
下游分为两个
途径,因此可以提供给用户两套不同的输出文档类
型:一类是提供源自正式的基因模式指定的蛋白质
和肽和
/
或者被鉴定的不同基因模式的证据;另外一
类是提供正式基因组注释在高度确信鉴定的位点
上有改善提高空间的证据。目前
ProteoAnnotator
目
前整合入
Proteosuite (
)
可
视化运行。
2.9 Galaxy-P
Galaxy-P
是基于
Galaxy
项目
(
ject.org)
的多
“
组学
”
数据特别关注于质谱为基础的
蛋白质组学的分析平台,在
Galaxy-P
的基础上,研
究人员开发出了一套高度灵活和宜使用的蛋白质
基因组学分析流程
(Jagtap et al., 2014)
。基于
Galaxy-P
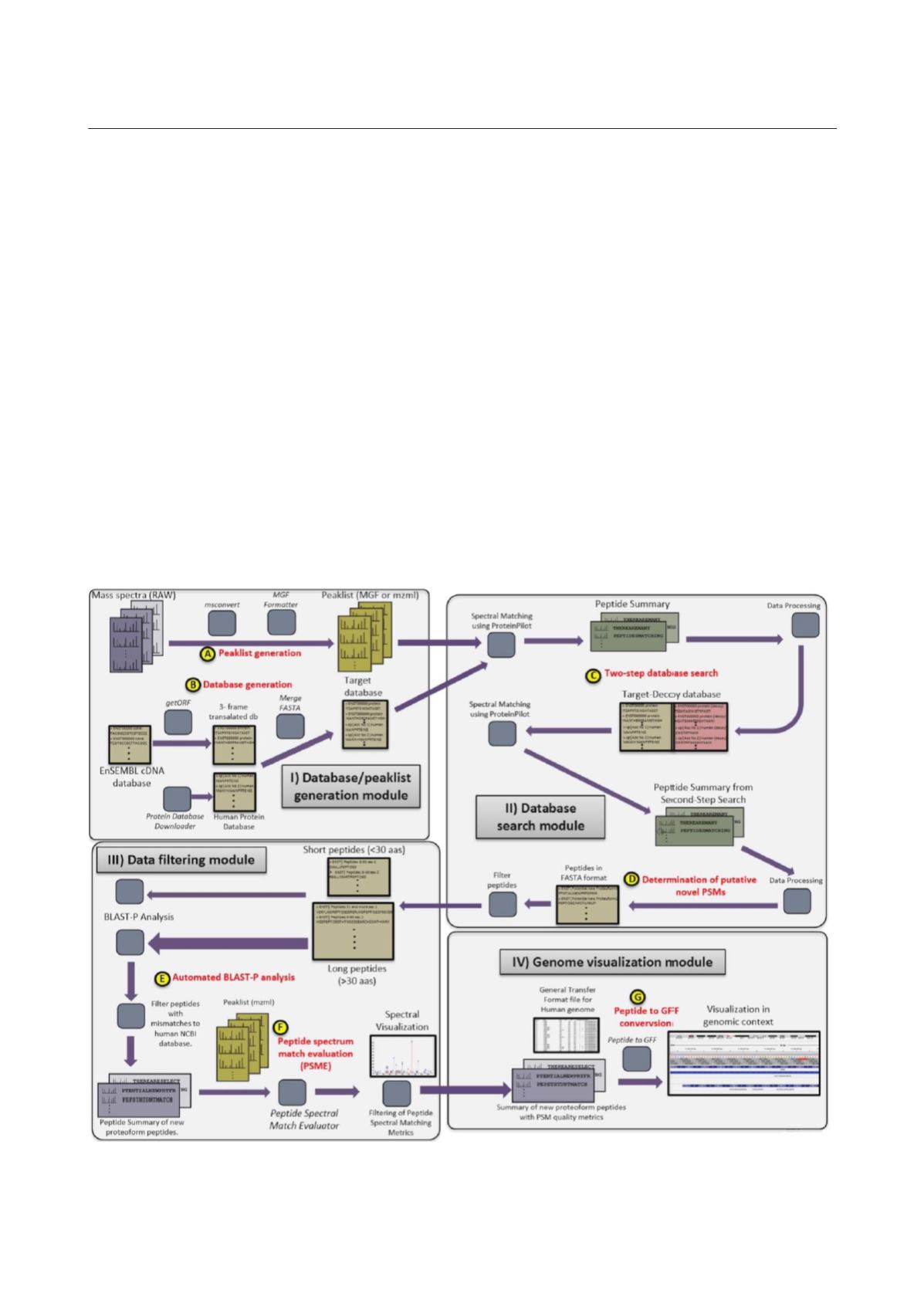
的蛋白质基因组学分析流程共包含大约
140
个处理步骤,可以归类为四个模块中,其具体
分析流程图如图
3
所示:
1
、质谱数据
Peaklist
文件
的生产和来自组装的
DNA
或
RNA
序列来源的蛋白
质序列数据库的生成;
2
、序列数据库的搜索;
3
、
数据过滤和可信度的分配;
4
、新蛋白质产物在基
因组中的可视化及阐释。
图
3 Galaxy-P
分析流程图
Figure 3 Overview of modules and subworkflows comprising the Galaxy-based proteogenomic analysis workflow


