Basic HTML Version

分子植物育种
(
网络版
)
Fenzi Zhiwu Yuzhong (Online)
1004
从上述实验结果中筛选牡丹
SRAP
的优化反
应体系为:总体积为
20 μL, Taq
聚合酶浓度为
1.000 U
;模板
DNA
浓度为
60 ng
;
Mg
2+
浓度为
1.500 mmol/L
;
dNTPs
浓度为
0.250 μmol/L;
引物
浓度为
0.500 μmol/L
。
1.3
‘凤丹’
SRAP-PCR
反应引物筛选
通过优化后的反应体系,对
500
对引物组合进
行扩增,从中筛选出
120
对多态性好,且谱带清晰
的引物组合用于后续实验,扩增结果
(
图
5)
。
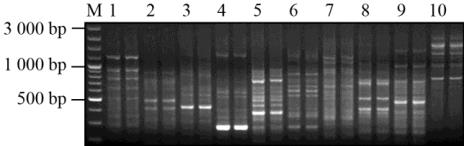
图
5 SRAP
引物筛选的扩增结果
(
引物
: Me23/ Em11
-
Em20)
注
: M: 100 bp Plus DNA Ladder; Me23:
正向引物
; 1~10:
Em11
-
Em20,
反向引物
,
每个处理重复
2
次
Figure 5 The amplification results of SRAP primers screening
(primers: Me23/ Em11
-
Em20)
Note: M: 100 bp Plus DNA Ladder; Me23: Forward primer;
1~10: Em11
-
Em20, Reverse primers, with 2 replicates
2
讨论
基于
PCR
反应开发的
SRAP
分子标记同样受到
反应条件和扩增程序变化及物种不同的影响,而利
用正交试验方法能考察各因素之间的相互效应,避
免单因素实验结果的不足,从而迅速获得满意的试
验结果
(
邹小云等
, 2010)
。因此,在构建牡丹遗传图
谱前进行‘凤丹’的
SRAP-PCR
反应体系的优化是
非常必要的。
我们在牡丹
SRAP-PCR
正交试验的基础上,分
别进行单因子优化试验,研究发现五个因素对
SRAP
扩增结果的影响具有差异,影响最显著的因
素是
Mg
2+
浓度,其次是引物浓度,这与苏美和和赵
兰勇
(2012)
的试验结果不同,推测可能是因为所用
牡丹材料的差异导致的结果。
我们结合单因子优化实验最终确定了‘凤丹’
的
SRAP-PCR
最佳反应体系为:反应体系总体积
20 μL,
含
10×PCR Buffer
,
Mg
2+
1.500 mmol/L
,
d
NTPs 0.250 μmol/L
、引物
0.500 μmol/L
、模板
DNA
60 ng
、
Taq
DNA
聚合酶
1.000 U
。利用优化后的
SRAP-PCR
反应体系,我们筛选出了
120
对多态性
好、谱带清晰的引物,这将为今后开展牡丹的遗传
多样性、油用和观赏牡丹遗传连锁图谱构建以及新
品种的鉴定等方面的研究奠定了基础。
3
材料与方法
3.1
供试材料与试剂
供试材料为保存于上海植物园科研中心牡丹
苗圃的江南牡丹品种‘凤丹’。实验用的
Taq DNA
聚合酶、
dNTPs
以及
100 bp DNA marker
购自
TaKaRa
公司,用于
SRAP
反应的引物等试剂均购
自上海生工生物工程技术服务有限公司,经初步筛
选,确定
Me2 (TGA GTC CAA ACC GGA GC)
和
Em10 (GAC TGC GTA CGA ATT CAG)
作为此次正
交试验的固定引物。
3.2
‘凤丹’基因组
DNA
提取
采用改良
CTAB
法提取‘凤丹’嫩叶的
DNA
,
琼脂糖凝胶电泳
(1.0%)
检测后,利用分光光度计
(NANODROP2000C)
检测基因组
DNA
的纯度和浓
度,最后将
DNA
稀释至
60 ng/μL
,保存在-
20
℃冰箱。
3.3
反应体系正交试验
采用正交设计
L
16
(4
5
)
进行试验,反应组合
(
表
2)
,并进行
3
次重复。
表
2 SRAP-PCR
反应的主要影响因子浓度梯度
Table 2 Factors and levels of SRAP-PCR reaction
水平
Level
Mg
2+
(mmol/L)
dNTPs
(mmol/L)
Taq
(
酶
)
Polymerase (U)
引物
(μmol/L)
Primers (μmol/L)
模板
DNA(ng)
Template DNA (ng)
1
1.500
0.100
0.500
0.200
30
2
2.000
0.150
1.000
0.300
60
3
2.500
0.200
1.500
0.400
90
4
3.000
0.250
2.000
0.500
120
分子植物育种
F
enzi Zhiwu Yuzhong

