Basic HTML Version


分子植物育种
(
网络版
), 2012
年
,
第
10
卷
,
第
1312
-
1316
页
Fenzi Zhiwu Yuzhong (Online), 2012, Vol.10, 1312
-
1316
http://mpb.5th.sophiapublisher.com
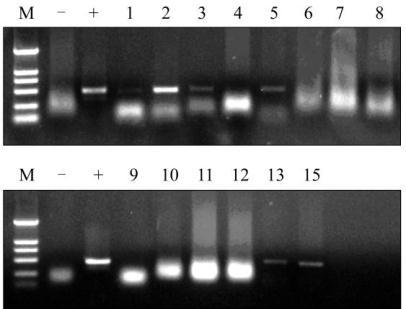
1315
图
5
棉花抗性植株
npt
Ⅱ扩增结果
注
: M: DL2000 DNA marker; 1~15:
抗性植株
;
-
:
未转基因
对照
; +:
阳性对照
Figure 5
npt
Ⅱ
amplification of cotton resistant plants
Note: M: DL2000 DNA marker; 1~15: Transgenic plant;
-
:
Non-transgenic plant; +: Positive control
否说明花粉管通道法的成功与否也与基因型依赖
有关,仍需要做进一步的实验分析。
3
材料与方法
3.1
试验材料和试剂
本实验用西和半夏无菌苗、大肠杆菌
(
E. coli
)
DH5α
、植物表达载体
pBI121
由本实验室保存。
Taq
DNA Polymerase
、
T
4
DNA
连接酶、限制性内切酶
购自大连宝生物公司,
Rnase
、
GEM-T vector
、
DNA
回收试剂盒等分别购自
Sigma
公司、
Promega
公司
和中科开瑞生物公司。
参照
Genbank
半夏凝集素基因
(AY191305.1)
的
核心序列,以及
Xba
Ⅰ和
Sma
Ⅰ酶切位点,设计了
PCR
扩增正、反向特异引物。引物由大连宝生物公
司合成。
以甘肃省棉花主栽品种陇棉
2
号
(
代号
k9505,
白棉
)
为实验材料。
3.2
半夏凝集素基因
(
pta
)
的扩增
按照
CTAB
法
(
王关林和方宏筠
, 1998)
提取半
夏模板
DNA
进行
PCR
扩增和产物回收,方法见参
考文献
(
张正英等
, 2010)
。
3.3
扩增产物的克隆及鉴定
依照说明将扩增的
PCR
产物克隆到
T-easy
载
体上,转化大肠杆菌
DH5α
。通过蓝白斑筛选、重
组质粒限制性内切酶双酶切和
PCR
扩增产物的电
泳结果进行鉴定。
3.4
序列测定与分析
阳性克隆送大连宝生物公司测序。测序结果用
BLAST
和
DNAMAN 6.0
软件分析。
3.5
植物表达载体的构建
按照张正英等
(2010)
叙述方法进行载体构建。
3.6
花粉管通道法介导的棉花遗传转化
参照《分子克隆》实验指南提取待转化质粒。
参照刘方等介绍方法
(
刘方等
, 2008,
中国棉花
, (8):
16)
进行转化,方法略有修改。先沿纵轴插入至子房
中轴约
2/3
处,然后再退至约
1/3
处开始注射。涂
抹赤霉素溶液
(
浓度
40 mg/kg)
于处理铃柄基部,以
减轻幼铃脱落。同时,对转基因铃绑线以区分非转
基因处理棉铃。质粒浓度
10~20 μg/mL
,每朵花注
射用量
10 μ
。
3.7
导入材料的田间筛选和室内分析
将收获的处理棉花种子种植于大田。利用
4 000~
5 000 mg/L
的卡那霉素在
T
1
棉花苗期、盛蕾期喷涂
倒
2
叶,选出抗性植株,单独收获。
将
T
2
棉铃种子按单粒种植,待长出第一片真叶
时进行
2 500 mg/L
的卡那霉素溶液涂抹,每天分早、
中、晚
3
次涂抹,连续
7 d
。统计棉花叶片的变化
情况。表现为抗性的材料进一步进行
4 000 mg/L
、
6 000 mg/L
的卡那霉素抗性检测。对检测到的抗性
植株做
PCR
检测。
作者贡献
张正英是项目负责人,指导实验设计和数据分析,完
成论文写作和修改。李淑洁、李静雯和王红梅是研究的执行
人,分别开展了部分试验研究,完成了相应的数据分析。
致谢
本文由基金项目甘肃省科技支撑项目
(1104WCGA188)
以及甘肃省农业生物技术应用与开发项目
(GNSW
-
2006
-
05)
资助完成。在项目实施过程中,得到了甘肃省农业科学院作
物所棉花课题组张秉贤研究员和冯克云副研究员的大力支
持,对此表示衷心感谢。
参考文献
Deng D.W., Guo S.D., and Yang Z.M., 1999, Study on the
molecular cytological mechanism of cotton transformation
by pollen tube pathway, Zhongguo Nongye Kexue
(Scientia Agricultura Sinica), 32(6): 113-114 (
邓德旺
,
郭
三堆
,
杨志民
, 1999,
棉花花粉管通道法转基因的分子
细胞学机理研究
,
中国农业科学
, 32(6): 113-114)
Guo S.D., Cui H.Z., Xia L.Q., Wu D.L., Ni W.C., Zhang Z.L.,

