Basic HTML Version


分子植物育种
(
网络版
), 2012
年
,
第
10
卷
,
第
1171
-
1178
页
Fenzi Zhiwu Yuzhong (Online), 2012, Vol.10, 1171
-
1178
http://mpb.5th.sophiapublisher.com
1175
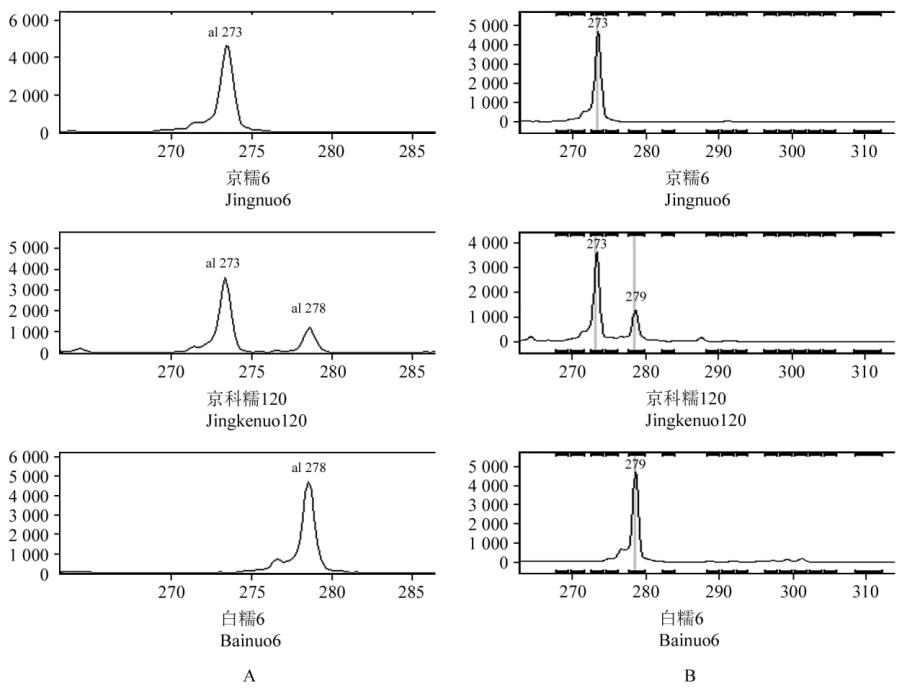
图
6
京科糯
120
及其亲本的荧光检测结果
(
引物
: umc1705w1)
注
: A: GeneMapper v3.7
分析结果
; B: GeneMarker-BMSTC v1.0
分析结果
Figure 6 PCR products of Jingkenuo120 and their parents detected with fluorescence detection (Primer: umc1705w1)
Note: A: The analysis result of GeneMapper v3.7; B: The analysis result of GeneMarker-BMSTC v1.0
图
7 GeneMarker-BMSTC v1.0
的过滤算法
Figure 7 The function of filter arithmetic of GeneMarker-BMSTC
v1.0
的分析数据吻合度达
99%
,说明两种软件可靠性都
非常高。同时
GeneMarker
相对于
GeneMapper
做出
了很多升级,比如二倍体筛选、自动去除
Pull-up
峰、识别连续峰、平移
Panel
等一系列功能,免除
了人工看板增删条带的繁杂工序,大幅度提高了数
据分析过程的自动化。
GeneMarker
软件因提供功能
订制,所以能够实现与数据库对接,即电泳后的
SSR
片段信息作为源数据,在软件自动分析及人工检查
后,数据可直接上传至指纹数据库系统,与数据库
的对接进一步完成了数据分析自动化的过程。
SSR
指纹数据分析不仅需要开发完善一个功能
强大的数据分析软件,还需要一些后续研究工作,比
如引物峰型特征描述、
BIN
的确定
(
王凤格等
, 2006)
。
SSR
标记具有种属特异性,不同农作物建立不同的
引物
Panel
,每对引物根据其等位基因特征建立
BIN
。
每对引物
BIN
的设置包括规定引物范围,各个等位
基因的名称、范围,以及记录等位基因的频率,标
注稀有等位基因等等。这些工作需要经过对大量样
品的分析,并且在
DNA
指纹数据库构建的过程中不
断累积完善,作为重要的备注信息添加到
SSR
分析

