Basic HTML Version


分子植物育种
(
网络版
), 2011
年
,
第
9
卷
,
第
1471
-
1478
页
Fenzi Zhiwu Yuzhong (Online), 2011, Vol.9, 1471
-
1478
http://mpb. chinese.sophiapublisher.com
1477
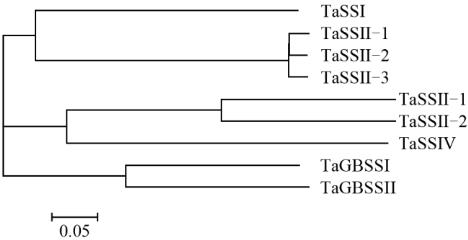
图
5
小麦淀粉合成酶进化关系
Figure 5 Phylogenetic relationships of wheat starch synthases
86% (Leterrier et al., 2008)
。每个亚家族中又可进一
步细化为单子叶植物和双子叶植物。
2
讨论
水稻和拟南芥基因组测序的完成推动了对其
基因和染色体的研究
(Yu et al., 2002)
,使得人们在
基因组水平上对植物重要基因家族进行挖掘、比较
分析和功能预测变为现实。特别是生物信息学相关
技术的开发,在植物大规模的基因分离、基因功能
注释及功能基因组学等各项研究中发挥越来越重
要的作用
(
王岩等
, 2009)
。普通小麦是一个异源六倍
体物种,有
A
、
B
、
D
三个不同的基因组。特别是
B
基因组含有丰富的抗病虫害等有益基因
(
于海霞和
田纪春
, 2008)
。对小麦各个染色体组进行有关起源、
进化、基因定位及基因与产量、生物性状的关联分
析,可更好的发掘和利用各染色体上的有利基因,
拓宽小麦的遗传变异基础,为在小麦育种中如何引
进新的遗传变异及杂优选育提供理论依据。
佃蔚敏等
(2004)
研究发现所有水稻
SS
基因家族
蛋白质的
C
-端氨基酸序列保守性均较强,和细菌的
糖原合成酶同源,与酵母和高等动物的糖原合成酶
同源性极低,并且认为植物淀粉合成酶与酵母和高
等动物的糖原合成酶可能源自于不同的祖先。小麦
基因组中存在九个编码淀粉合成酶的基因,分散在
不同染色体或染色体的不同位点,非成簇分布。和
小麦相似,拟南芥和水稻基因组中淀粉合成酶也分
别由不同位点基因编码,说明淀粉合成酶基因家族
可能是由染色体大片断倍增形成的
(
佃蔚敏
, 2004)
。
水稻基因组中淀粉合成酶基因的倍增可能发生在
单子叶和双子叶植物分化之后,禾本科植物分化之
前。小麦、玉米、高粱和大麦等禾本科植物基因组
中淀粉合成酶基因也发现有类似的的倍增
(
佃蔚敏
,
2004)
。
目前,关于植物淀粉合成酶基因的分子起源和
进化研究较少。然而,研究基因的倍增方式、分化
途径可以预测该基因在自然选择的压力下进化过
程中的命运
(Sankoff, 2001)
,这些研究将有利于对植
物淀粉合成酶基因的表达特性、调控机理和生理功
能的进一步探索,同时可为深入研究小麦淀粉合成
的分子机理和品质的遗传改良提供理论基础。
3
试验材料与方法
3.1
试验材料
从
NCBI (http://www.ncbi.nlm.nih.gov)
最新数据
库下载六倍体小麦相关淀粉合成酶核酸和氨基酸
序列,以及其它相关作物的淀粉合成酶氨基酸序列。
3.2
淀粉合成酶基因系统发生学分析
应用多序列比对工具
Clustal X
构建系统进化树
(Ramu et al., 2003)
,以相关淀粉合成酶氨基酸全序
列联配的结果为基础,用
MEGA 4.0
程序生成
(Tamura et al., 2007)
。采用程序中的
Neighbor-joining
法,运行采用如下参数:选择
Poisson Correction(
泊
松修正
)
这一模型,缺口设置为“
Pairwise Deletion
”,
节点支持率使用
1 000
次
Bootstrap
检验。
3.3
淀粉合成酶氨基酸序列分析
通过
Pfam
网站
(The protein families database,
http://www.sanger.ac.uk/Software/Pfam/)
,根据淀粉合
成酶结构的不同,将其进行分类。通过
Pfam
数据库
进行系统分析得到的淀粉合成酶蛋白质结构域表
现形式,进一步分析不同结构域的各氨基酸频率。
3.4
淀粉合成酶基因结构分析
通过
Gene Structure Display Server
在线基因结
构分析工具,分析部分淀粉合成酶基因全长的结构。
作者贡献
李春艳、李诚是本研究的实验设计和数据分析的执行
人,并完成论文初稿的写作;李卫华、曹连莆指导实验设计
与论文修改。全体作者都阅读并同意最终的文本。
致谢
本研究由石河子大学高层次人才科研启动资金专项
(RCZX201004)
和石河子大学科学技术研究发展计划动植物
育种专项
(GXJS2008
-
YZ04)
共同资助。本文中提到了我们研究

