基本HTML版本
昆虫分子生物学研究
(
网络版
), 2013
年
,
第
2
卷
,
第
1-4
页
Kunchong Fenzi Shengwuxue Yanjiu (Online), 2013, Vol.2, 1-4
http://imbr.biopublisher.cn
2
小菜蛾是以十字花科植物为食的昆虫,由于其对重
要的十字花科作物带来毁灭性的危害,以及对化学
农药及生物农药产生抗药性,成为鳞翅目昆虫中最
引人关注的农业害虫之一
(Talekar N.S., & Shelton
A.M., 1993; Furlong et al., 2012)
随着现代生物技术的迅速发展,在分子水平上
研究小菜蛾生物学特性成为研究热点,在基因组
学、分子遗传、分子进化以及分子适应方面取得了
显著的进展
(Xie, 2013)
。
1
小菜蛾的基因组
小菜蛾的基因组大小约为
343 Mb
,预测含有
18,071
个蛋白编码蛋白,
781
个非编码
RNA
,基因
组中有
33.97%
重复序列组成
(You et al., 2013)
。小菜
蛾拥有
1,412
个特有基因,这些特有基因大都参与
与环境信息处理、染色体复制或修复、转录调节以
及碳水化合物、蛋白质代谢等基本的生物学途径
(You et al., 2013)
。
2
小菜蛾的分子遗传
小菜蛾自身高度杂合,有
31
条染色体(
n=31
),
存在丰富的
SNP
、插入缺失、结构变异以及复合片
段重复等
DNA
变异(图
1
)(
You et al., 2013
)。
Baxter
et al.
利用下一代
RAD
测序技术构建了小菜蛾的连
锁图谱
(Baxter et al., 2011B)
。将
3,177
个母校遗传
的
RAD
归属到
31
个染色体上,使得能够鉴定农药
抗性和
W/Z
性染色体;用来自回交父本的
2,878
个
分离的等位基因座构建了
1292 cM
长的基因组连锁
图(图
2
)。同年,
Baxter
还报道了一个膜转运子
(ABCC2)
的基因座遗传定位,这个基因座控制着两
个鳞翅目昆虫的
Cry1Ac
毒蛋白抗性,说明这个蛋
白在
Bt
功能上起着关键的作用
(Baxter et al.,
2011A)
。
3
小菜蛾的分子进化
小菜蛾是动物界、节肢动物门、昆虫纲、鳞翅
目、菜蛾科、菜蛾属一种昆虫。小菜蛾大约在
124
万年以前与其它两个鳞翅目昆虫,家蚕(
B. mori
)
和黑脈金斑蝶(
D. plexippus
)中分化出来。利用现
有昆虫基因组信息构建系统发育树证实小菜蛾属
于鳞翅目昆虫中一个原始的类型(
You et al., 2013
)。
系统发生分析确认昆虫目的分化时间大概在
265~332
万年以前,这与单子双子叶植物分化时间
为
304
万年以前相一致,证实了昆虫行为与宿主植
物存在共进化。当十字花科植物从番木瓜科
caricaceae
分化出来时(
54~90
万年以前),小菜蛾
即成为十字花科植物专食昆虫。
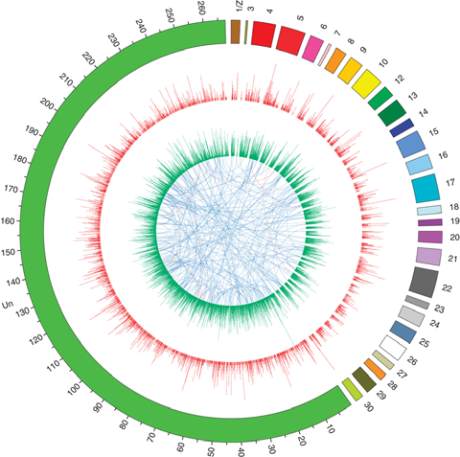
图
1
测序小菜蛾(
P. xylostella
)中的基因组变异
(
摘自:
You
et al., 2013)
注:最外面的圆圈表示用
100 kb
单位尺度装配的参照基因
组
;
能被指派到连锁群的
Scaffolds
以随机顺序组合产生
28
个染色体的部分序列
;
绿色的部分表示这些
Scaffolds
不能
被指派
(Un);
最里面的圆环表示大于
8kb
的片段重复,连接
线表示原始片段和副本位置
; 100%
相似性的片段重复用红
色表示,大于
90%
相似性的片段用蓝色表示
;
柱状图则分别
表示单核苷多态性
(SNPs)
的数量
(
红色
,
外圆
)
以及在
30 kb
和
50 kb
范围中的插入和缺失
(Indels)(
浅绿色
,
内圆
)
Figure 1 Genomic variations within the sequenced
P. xylostella
strain (Adopted from You et al. 2013)
Note: The outermost circle shows the reference genome
assembly with a 100-kb unit scale. Scaffolds that could be
assigned to linkage groups are joined in arbitrary order to
generate the partial sequences of 28 chromosomes.The green
segment represents the scaffolds that were unable to be
assigned (Un). The innermost circle denotes segmental
duplications (of≥8 kb), with connections shown between
segment origins and duplication locations. Segmental
duplication pairs with 100% similarity are shown in red, and
those with ≥90% similarity are shown in blue. Histograms
indicate the number of SNPs (red, outer circle) and indels (light
green, inner circle) in 30-kb and 50-kb windows, respectively

